GedMatch
GEDmatch ist eine kostenlose Analyseplattform für genealogische Daten, auf der man die Ergebnisse seiner DNA-Analyse mit anderen Nutzern vergleichen ('Matching') kann. Ausserdem sind Ethnizitätsschätzungen bzw. Herkunftsanalysen ('admixture') möglich. Darüber hinaus gibt es weitere Werkzeuge, wie z.B. 'Phasing' und einen 3D-Chromosomen-Browser.
Eine Anleitung zur Arbeit mit GEDmatch
Die ersten Schritte
Ein Konto erstellen
Auf der Seite www.gedmatch.com unten auf der Seiten auf die Frage "Not registered?" Click here dem Link folgen. Die nächste Seite trägt den Titel "User registration". Dort bitte wie folgt vorgehen:
a) unter „First & Last name“ bitte den jeweiligen Vor- und Familiennamen eingeben
b) unter "Alias" kann ein Pseudonym gewählt werden
c) Danach bitte zweimal dieselbe E-Mail-Adresse eingeben. Diese Adresse erscheint später im Benutzer-Register und auf der Seite mit den Treffern. Vorsichtige benutzen hier manchmal gern eine gesonderte E-Mail-Adresse im Kontakt mit ihren DNA-Treffern. An diese Adresse wird nach Abschluss der Registrierung ein Code geschickt, also sollte das E-Mail-Konto durchaus funktional und zugänglich sein.
d) Das Passwort ebenfalls zweimal eingeben. Es sollte mindestens 6 Zeichen enthalten, darunter eine Zahl und möglichst eine Zahl oder ein Symbol.
e) Mit "Register" die Anmeldung abschließen.
Es erscheint ein Bildschirm mit einer weißen Freifläche. In diese Fläche bitte nun den roten "Registration Confirmation Code" eintragen, der per E-Mail versandt wurde. Mit "confirm" abschließen.
Rohdaten hochladen
Bevor Rohdaten zu GEDmatch hochgeladen werden können, sollten sie auf dem eigenen Computer vorhanden sein. (Anmerkung: Auf GEDmatch selbst gibt es auf Englisch unter "Generic Upload Fast" eine genaue Anleitung für den Download der Daten von den großen Testanbietern (Übersetzungen folgen).
Dazu bitte zunächst die Rohdaten ("Raw Data") der Testfirma herunterladen, in den meisten Fällen gibt es eine Auswahlmöglichkeit "download raw data". Kunden von FTDNA sollten die Schaltfläche "Build 36 Raw Data Concatenated" zum Download (Herunterladen) nutzen.
Es ist empfehlenswert, sich den Speicherort der Datei zu merken bzw. auch den Namen der heruntergeladenen Datei zu notieren, um sie später auch lokalisieren zu können. Die Datei sollte im heruntergeladenen Zustand nicht entpackt („unzip“) werden, da sie im nächsten Schritt in genau diesem Zustand zu GEDmatch hochgeladen wird.
Nachdem die Rohdaten nun auf dem eigenen Computer gespeichert sind, kann das Hochladen (Upload) erfolgen.
Dazu bitte bei GEDmatch anmelden (Login) und auf der rechten Seite den Punkt "Generic Upload Fast" wählen. Es öffnet sich das sogenannte "GEDmatch raw DNA upload utility", also das Werkzeug, mit dem die Rohdaten der verschiedenen Anbieter zu GEDmatch hochgeladen werden können.
Diese Seite bitte wie folgt ausfüllen:
1. Das erste weiße Eingabefeld mit dem Namen derjenigen Person füllen, zu der diese DNA-Probe gehört. Wer seinen richtigen Namen nicht in der Datenbank wiederfinden will, wählt beim nächsten Feld ein Pseudonym.
2. Dieses Feld kann ein Pseudonym enthalten oder frei bleiben.
3. Bei der Auswahlmöglichkeit "male" (männlich) bzw. "female" (weiblich) bitte das Geschlecht der getesteten Person eingeben.
4. Unter "What is your mitochondrial Haplogroup?" bitte die Haplogruppe aus einer eventuellen mtDNA-Analyse angeben, ansonsten unausgefüllt lassen.
5. Das nächste Feld fragt nach der Haplogruppe der yDNA, das kann die eigene sein (bei einem männlichen Teilnehmer) oder die einer anderen getesten männlichen Person (Bruder, Onkel etc), zu der die Probe gehört.
6. Der nächste Punkt beinhaltet die Zustimmung zur Veröffentlichung der Daten zu Vergleichszwecken in der Datenbank. Wird dieser Punkt nicht bejaht, können keine Daten verglichen, sprich "Matches" gefunden werden.
7. Anschließend bitte die Schaltfläche "Browse" wählen, um die zuvor gespeicherten Rohdaten auf dem eigenen Computer zu finden.
8. Mit Upload dann die ausgewählten Daten hochladen.
9. Jetzt ist es wichtig, Geduld zu haben. Während die Daten hoch geladen werden, bitte warten und den Ladevorgang nicht durch Schließen des Bildschirms beenden. Normalerweise sieht man das Hochladen der einzelnen Chromosomen. Der gesamte Prozess kann einige Minuten in Anspruch nehmen, idealerweise wählt man von Europa aus eine Nachtzeit in den USA, besonders bei langsamer eigener Internetverbindung.
Warten
Nachdem die Rohdaten hochgeladen sind, beginnt GEDmatch, diese mit der gesamten Datenbank abzugleichen. Dieser Prozess kann einige Tage dauern. Bis zu diesem Zeitpunkt können aber schon einige Werkzeuge benutzt werden, z. B. die Ethnizitätsschätzungen unter "Admixture" oder der 1:1 – Vergleich unter "One-to-One". In der Regel dauert es ein bis mehrere Tage, bis alle frei zugänglichen Funktionen benutzt werden können.
Der one-to-many-Vergleich
In englischer Sprache sind folgende Anleitungen für GEDmatch empfehlenswert:
- Jared Smit: "Genetic Genealogy using GEDmatch. An Absolute Beginners Guide".
- Louise Coakley: Tips for using GEDmatch. Blog Genie1.
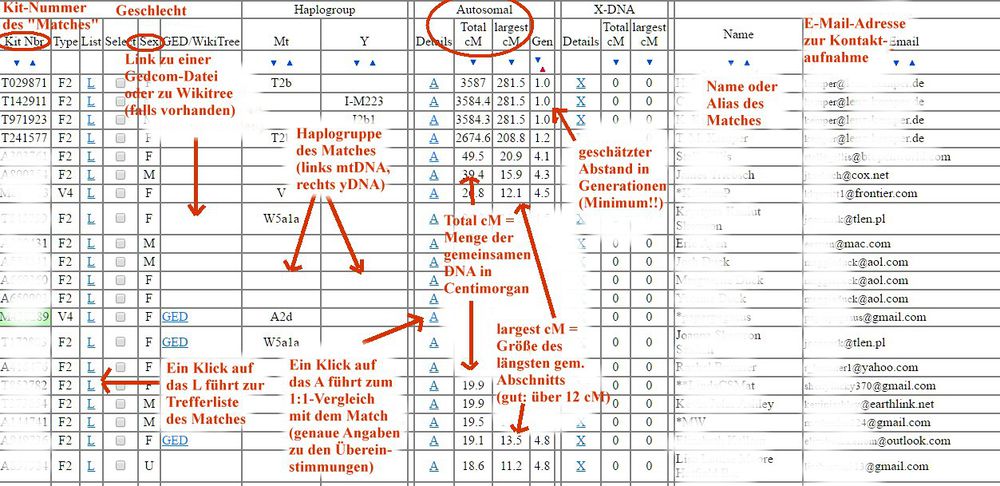
Für deutschsprachige Nutzer mag vorläufig folgende Grafik eine Hilfe sein:
Der 1:1-Vergleich ("One-to-one")
Dieses Werkzeug dient in erster Linie dazu, eine Verbindung, die man dem Vergleich mit der gesamten Datenbank (also im "One-to-many"-Vergleich) entdeckt hat, genauer anzuschauen. GEDmatch empfiehlt, auf jeden Fall zumindest mit diesem direkten Vergleich zu testen, bevor man Kontakt zu einem der Treffer ("Matches") aufnimmt.
Wenn man die Funktion öffnet, gibt es mehrere freie Felder:
In die ersten beiden Felder trägt man die GEDmatch-Nummern der zu vergleichenden Datensätze ein. ("Kit no. 1" und "Kit no. 2"). Es folgt eine Auswahl der möglichen Darstellungsformen:
1. Graphische Darstellung mit Start- und Endpositionen der gemeinsamen Segmente, CentiMorgan und SNPs ("Graphics and Positions")
2. Darstellung der gemeinsamen Segmente in Tabellenform ("Position only"), diese Option ist voreingestellt und bringt bei langsamer Internetverbindung die schnellsten Ergebnisse
3. Rein graphische Darstellung ("Graphics only")
Im unteren Teil kann man nun die Parameter setzen. Für den Anfänger empfiehlt es sich, die Voreinstellungen zu übernehmen und mit "submit" ganz unten auf der Seite das Ergebnis aufzurufen.
Ansonsten gibt es folgende Änderungsmöglichkeiten:
Die Bildqualität kann mit der Auswahl "Full resolution" oder der Eingabe der entsprechenden Pixelzahl variiert werden oder bleibt im voreingestellten Modus.
Im nächsten Schritt können die Grenzwerte für SNPs (Standardeinstellung ist 500 SNPs) und Centimorgan (Standardeinstellung bei 7 cM als Grenze für das kleinste angezeigte Segment) verändert werden.
Wenn die Felder nicht ausgefüllt werden, greift das Programm auf die Standardeinstellung zurück. Diese sollte man zunächst bei wenig Erfahrung oder für einen ersten Schnell-Check beibehalten, um eine möglichst hohe Sicherheit zu haben, dass die angezeigten Segmente auch tatsächlich einem gemeinsamen Vorfahrenpaar entstammen und nicht zufällig (IBS) sind. Bei bekannten und/oder gesicherten Verwandtschaftsverhältnissen kann man dann beginnen, sich auch die kleineren Segmente z. B. mit 5 cM und 500 SNPs anzuschauen.
Mit der Schaltfläche "submit" gelangt man zu den Resultaten des Vergleichs.
Belässt man es bei den voreingestellten Werten, enthält das Ergebnis je nach Auswahl graphisch, in Tabellenform oder in Kombination der beiden die übereinstimmenden Segmente sowie eine Schätzung des Verwandtschaftsverhältnisse im Textteil am Ende der Vergleichsseite.
Die Ergebnisseite kann so aussehen: 1. Der Treffer ist ein Treffer:
Comparing Kit T00000 (*Name A) and T000001 (Name B) Vergleich Kit T.... (*Name) mit T... (*Name) Chr Start Location End Location Centimorgans (cM) SNPs 19 13,935,959 18,513,521 9.8 1,383 Largest segment = 9.8 cM (größtes Segment = 9.8 centiMorgan) Total of segments > 7 cM = 9.8 cM (Gesamtzahl Segmente größer/ länger als 7 cM = 9.8 cM) 1 matching segments (ein übereinstimmendes Segment) Estimated number of generations to MRCA = 5.5 (geschätzte Anzahl der Generationen bis zum nächsten gemeinsamen Vorfahren = 5.5)
Anmerkung: Die geschätzte Generationenanzahl hängt natürlich von verschiedenen Faktoren ab (Endogamie, Eheschließungen unter Cousins, außerdem kann natürlich der Abstand der beiden Probanden zu diesem gemeinsamen Vorfahren auch sehr unterschiedlich sein) und sollte daher eher als grobe Schätzung verstanden werden.
2. Der Treffer ist kein Treffer oder nur bei sehr kurzen Segmenten/ wenigen SNPs:
Comparing Kit T0000000000(Name) and T0000002 (Name) (Vergleich von Kit T... und T...) Minimum threshold size to be included in total = 500 SNPs (Verwendeter Grenzwert für SNPs = 500 SNPs mindestens) Mismatch-bunching Limit = 250 SNPs Minimum segment cM to be included in total = 7.0 cM (Verwendete Mindestlänge cM = 7.0) Largest segment = 0.0 cM (längstes Segment) Total of segments > 7 cM = 0.0 cM (Anzahl der Segmente über 7 centiMorgan = 0.0 cM) (2228) No shared DNA segments found (Keine gemeinsame DNA gefunden) 676112 SNPs used for this comparison. (Für den Vergleich wurden 676112 SNPs verwendet.) Comparison took 0.04726 seconds. (Der Vergleich dauerte 0,04276 Sekunden.)
Siehe auch
Weblinks
- www.gedmatch.com Website von GEDmatch
- GEDmatch Hilfe auf ISOOG (engl.)
- Wohin mit den Rohdaten? Zu GEDmatch! Deutschsprachiger Blogbeitrag von Anja Klein auf 'Welt der Vorfahren'.
- Videos auf youtube Einführungen in englischer Sprache
- Genealogical Musings Finally! A Gedmatch Admixture Guide! Blog
- Using GEDMatch(pdf) Anleitung (engl.).
Anmerkungen

|